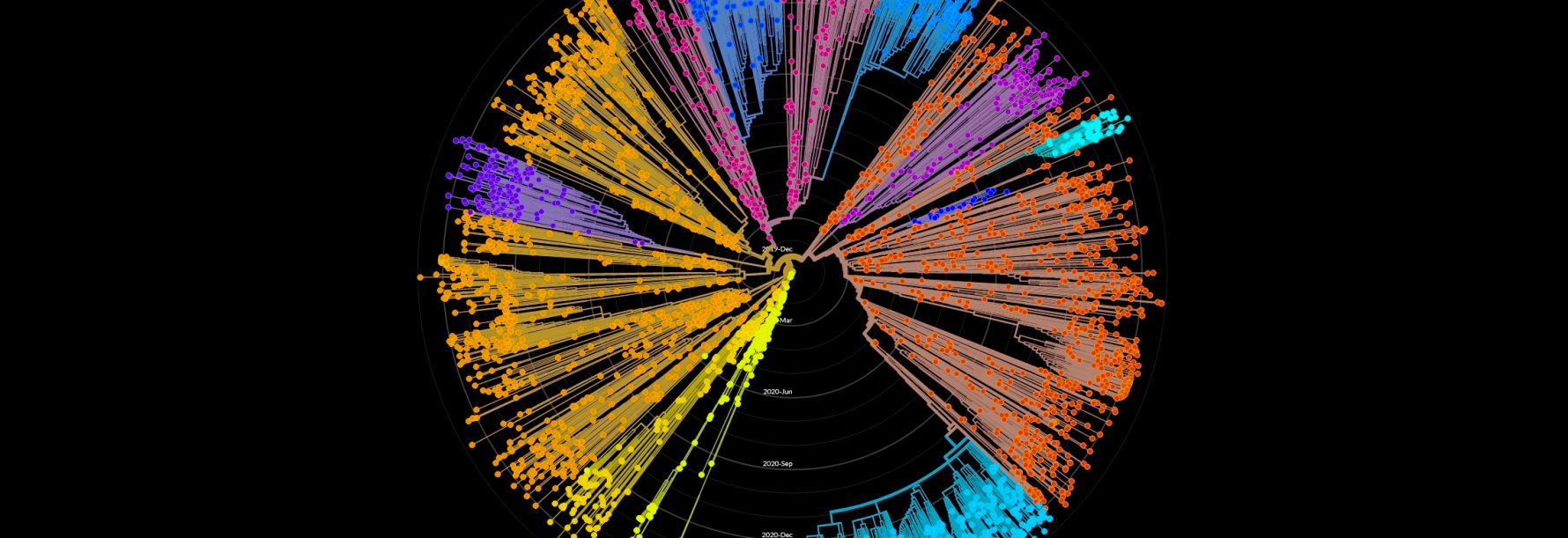
Inzicht in ontstaan van leven, in samenspel met de omgeving, is een van de belangrijkste wetenschappelijke ontwikkelingen van de laatste 200 jaar. Het vervolg daarop is het voorspellen van evolutionaire processen. Evolutiebiologie gaat een belangrijke rol spelen bij het oplossen van enkele van de grote uitdagingen waarmee we vandaag de dag worden geconfronteerd. De ontwikkeling van bacteriële resistentie, resistentie tegen pesticiden, uitbreken van nieuwe ziekten, en de aanpassing van soorten aan de verstedelijking en klimaatsverandering. Het zal ook eenvoudiger worden biodiversiteit te bevorderen.
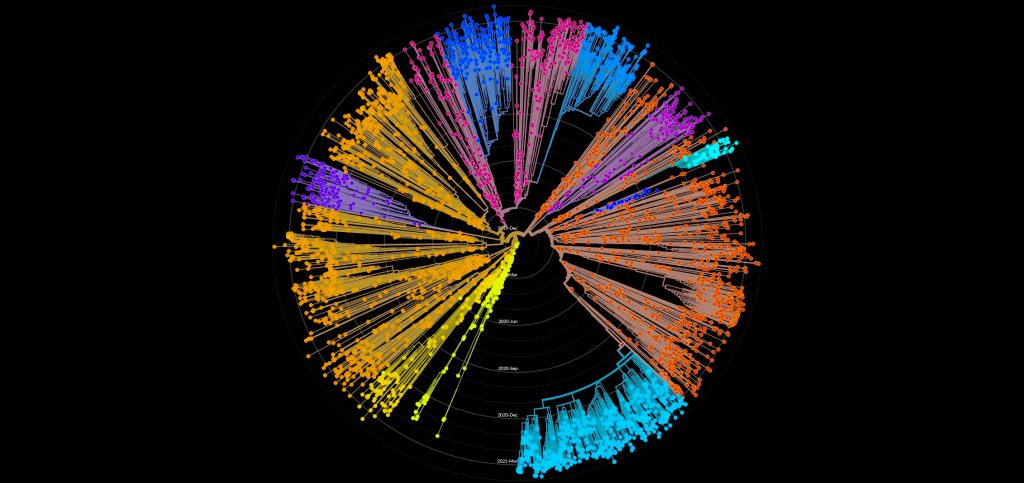
Onderzoekers werken aan twee voorwaarden om evolutie te voorspellen.
1. Onderzoekers moeten inzicht krijgen welke processen precies plaatsvinden tijdens evolutie, op verschillende niveaus van hiërarchie en schaal. Dat betekent dat evolutie van een soort afhankelijk is van andere soorten in dezelfde voedselketen en hetzelfde ecosysteem. Maar ook van (bio)moleculen tot cellen, van organismen tot ecosystemen tot de hele biosfeer van onze planeet. De echte doorbraak zal bestaan uit het voorspellen van de evolutie van de huidige soorten die zich de komende jaren aanpassen aan hun veranderende omgeving. Doe je met ons mee?
2. Ook brengen onderzoekers goed in kaart welke veranderingen we op onze planeet verwachten. Als we goed inzicht hebben in veranderingen van het klimaat, het landschap en de oceanen, is het mogelijk te voorspellen welke effecten dit zal hebben op de evolutie van dieren en planten op aarde.
Kennisnetwerk voorzitters

Bregje Wertheim
Member Steering Committee
University of Groningen

Bregje Wertheim
Member Steering Committee
University of Groningen
Networks:
Fields of interest:
I am fascinated by the genomic basis of evolution: how the genome shapes and affects the ability of organisms to adapt. It is only since a decade that technology allowed us to start quantifying genomic variation within and among species. In my research, I have been employing state-of-the-art technologies and analyses to investigate the true complexity of evolutionary processes and answer long-standing questions in evolution, mapping how genomes change during evolution and how these changes are manifested in gene networks.

Liedewij Laan
Member Steering Committee
TU Delft

Liedewij Laan
Member Steering Committee
TU Delft
Networks:
Fields of interest:
As pioneers of the emerging field of evolutionary cell biophysics, we aim to understand how the building blocks of a cell constrain and facilitate evolution of cellular functions. The function we focus on is symmetry breaking in budding yeast. We do experimental evolution, quantitative cell biology and modeling in live cells in combination with minimal in vitro systems to understand the molecular mechanisms of adaptive mutations and to predict fitness, both with bottom-up (biophysics) and top-down (statistical) approaches.
Sluit je aan bij deze netwerkgroep
Als subgroep organiseren we een reeks workshops voor de Nederlandse evolutiegemeenschap. Het doel van deze workshopserie is om elkaar technieken en “talen” te leren die in verschillende subvelden van de evolutie worden gebruikt, om toekomstige samenwerkingsverbanden te faciliteren.
De opzet is als volgt: twee sprekers geven een 15-20 minuten durende (online) lezing over een specifiek onderwerp. Na de lezingen zijn er 45 minuten voor vragen/discussies of om bijvoorbeeld aan een kleine opdracht te werken.
Het is goed om te weten dat dit educatieve lezingen zijn in plaats van seminars met de nieuwste en coolste resultaten.
PS: Wij stellen suggesties voor onderwerpen/sprekers zeer op prijs.
Wanneer: Derde dinsdag van de maand om 16:00 uur.
Waar: op Zoom
Wie: Voor iedereen die zich deel voelt van de Nederlandse Evolutie gemeenschap en die wil leren! (Doctoraten / Post-Docs / Staf)
Hoe? Schrijf je hier in voor de lezingen of voor de mailinglijst*. Een dag voor de workshop ontvangt u een e-mail met een Zoom-link en een wachtwoord.
Contact: Bregje Wertheim of Liedewij Laan.
* Als u zich inschrijft voor het gratis lidmaatschap van het Origins Center, ontvangt u automatisch mails van de kennisnetwerken die u in het inschrijvingsformulier hebt geselecteerd.